VBMEG Tutorial
VBMEG Tutorial
はじめに
VBMEGは、MEGやEEGのデータから皮質電流を推定するためのMatlabツールボックスである。 本チュートリアルは、サンプルデータの解析を通じて、VBMEGの処理の流れを把握することを目的としている。
準備するファイル
http://vbmeg.atr.jp/?lang=ja#DOWNLOADよりダウンロードできます。
- MEGデータファイル(A008a_BC_0.5HPF_100LPF_trig437_label4.ave)
- センサ位置合わせファイル(marker1.pos.mat)
- MRI構造画像ファイル(3D.hdr, 3D.img)
- 脳モデルファイル(lh.(curv/inflated/smoothwm).asc, rh.(curv.inflated/smoothwm).asc)
MEGデータファイル
- 横河電機MEGデータファイル
- 聴覚刺激課題
- 3.2kHzの純音を、左耳に提示したときの脳活動を加算平均したもの。
センサ位置合わせファイルの詳細
- MEGのセンサ座標を被験者のMRI構造画像座標系に位置合わせするための情報が入ったファイル。
- 位置合わせプログラムにより作成する。
MRI構造画像ファイルの詳細
- Analyze 7.5形式。中のデータ並びは左手系(LAS)。
以下のHPが参考になる。
http://www.wideman-one.com/gw/brain/orientation/orientterms.htm
脳モデルファイルの詳細
- FreeSurfer(http://surfer.nmr.mgh.harvard.edu/)から作成した個人脳ファイル。
- MRI構造画像ファイルから作成することができる。
ディレクトリ
- 以下のようにプログラムおよび入力データが格納されていることを前提にしている。
D:\vbmeg(VBMEGプログラムディレクトリ)
D:\data(入力データディレクトリ)
│ 3D.hdr
│ 3D.img
│
├───FS
│ lh.curv.asc
│ lh.inflated.asc
│ lh.smoothwm.asc
│ rh.curv.asc
│ rh.inflated.asc
│ rh.smoothwm.asc
│
└───Yokogawa
A008a_BC_0.5HPF_100LPF_trig437_label4.ave
marker1.pos.mat
作業手順
パスを設定する
- MATLABを起動し、実行準備をする。

プロジェクトを作成する
- コマンドラインからproject_mgrを起動する。
>> project_mgr
- [File]->[New project]を選択する。
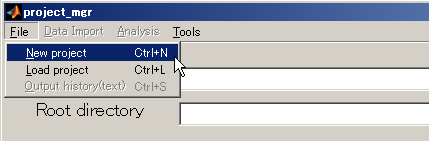
- プロジェクト名を入力し、Selectボタンを押す。
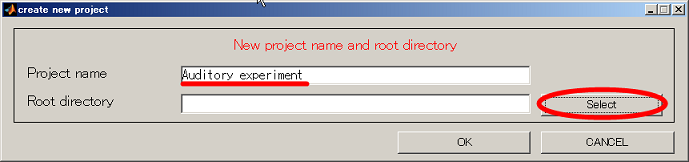
- カレントディレクトリをD:\に変更し、make dirボタンを押す。表示されたダイアログにAuditoryと入力し、OKボタンを押す。
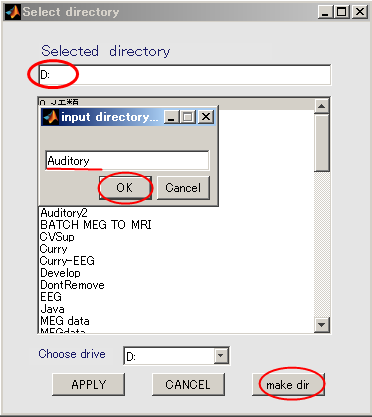
- 作業ディレクトリ(D:\Auditory)を選択し、APPLYボタンを押す。
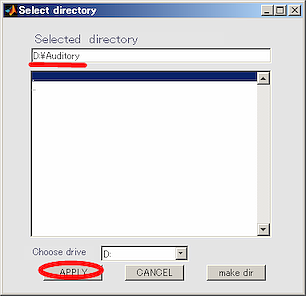
- OKボタンを押し、ダイアログを閉じる。
- 確認ダイアログが出るので、「はい」を選択する。
- project_mgrの準備が整った。
脳モデルをインポートする
- [Data Import]->[Import cortical model]を選択する。
- MRI構造画像ファイル, 脳モデルファイルをセットする(最初にラジオボタン:FreeSurferを押し、各Selectボタンを押して、ファイルを選択する)。
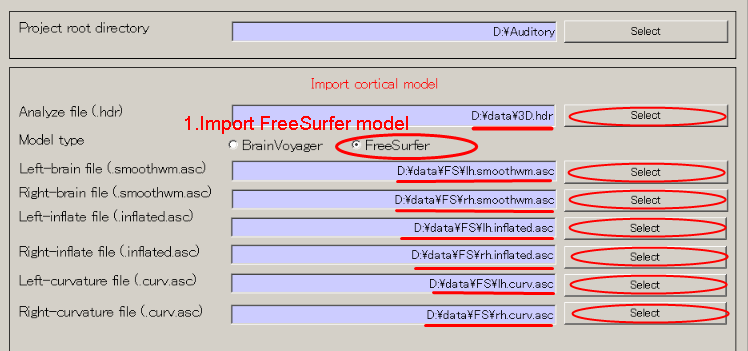
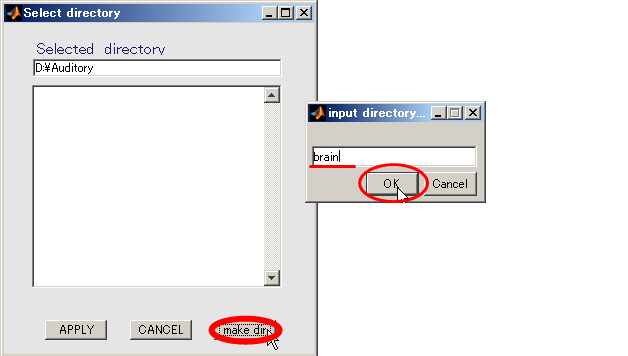
- 出力先を作成する(D:\Auditory\brain)
- Selectボタンを押す。

- make dirボタンを押すと、作成するディレクトリ名を入力するダイアログが表示されるので、brainと入力し、OKボタンを押す。
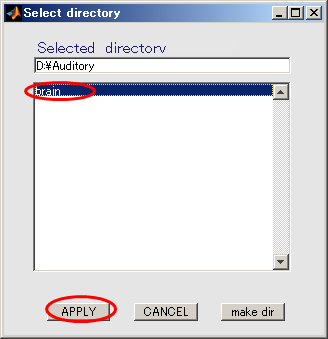
- brainを選択してから、APPLYを押す。
- 保存ファイル名が更新される。
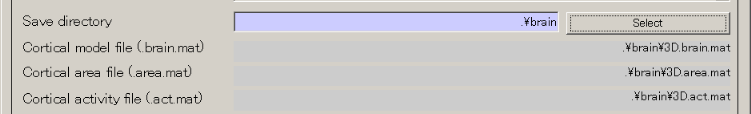
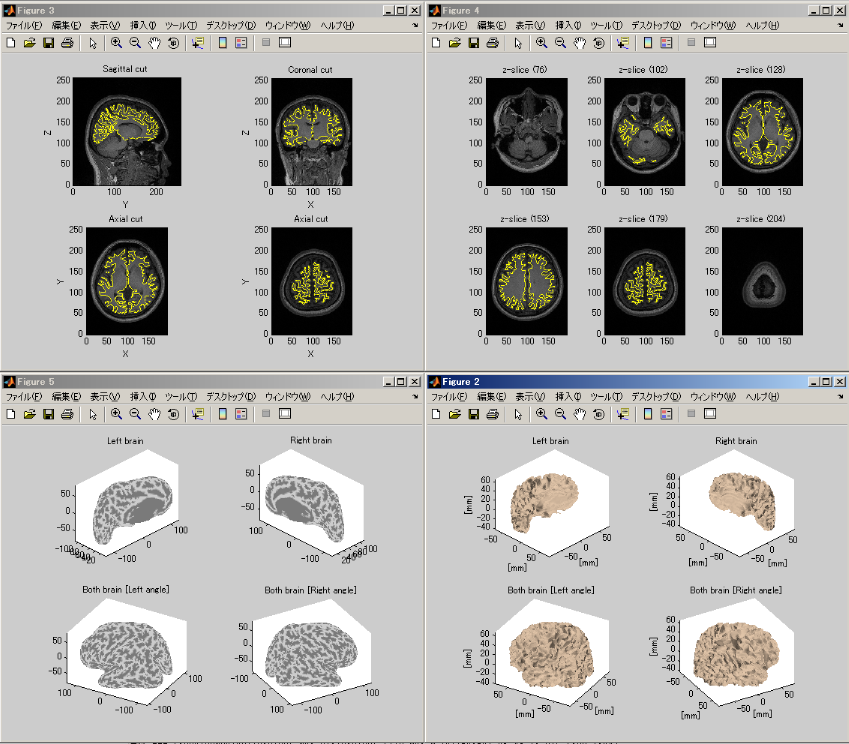
- Execボタンを押して、20分ぐらい待つと、脳モデルのインポートが完了し、結果が表示される。
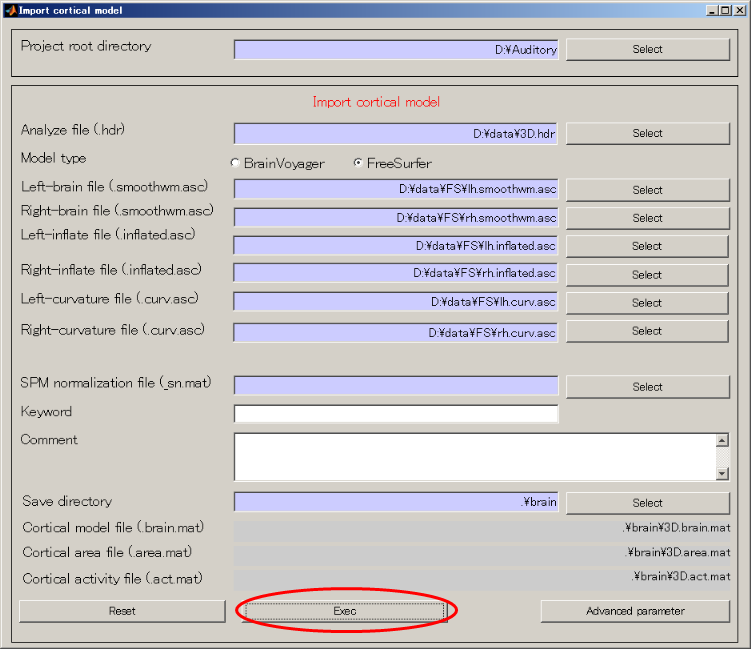
結果画面
MEGデータをインポートする
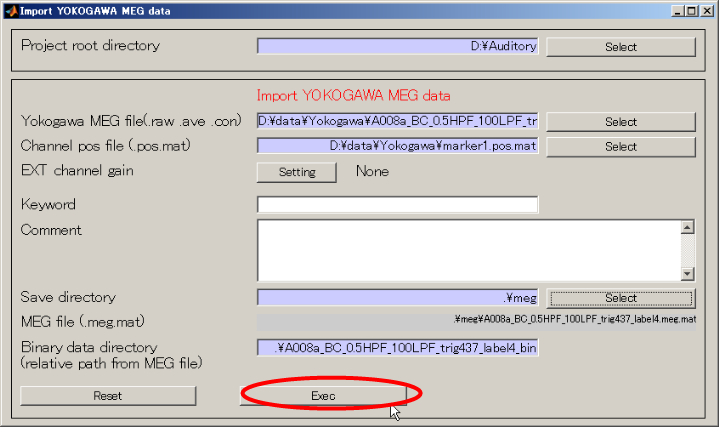
- [Data Import]->[Import MEG data]->[Yokogawa]を選択する。
- Yokogawa MEG fileを指定する。
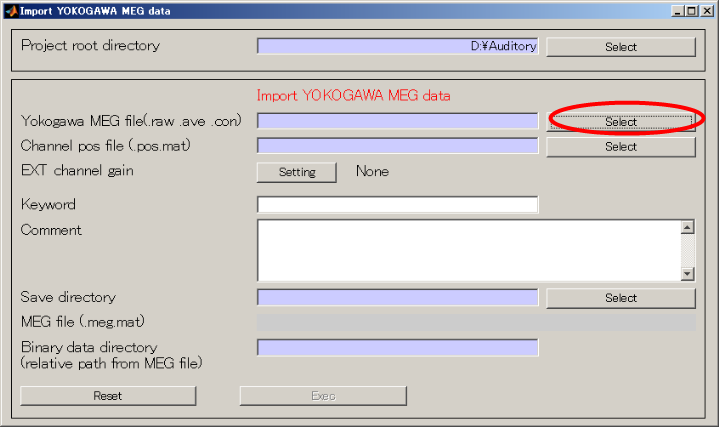
- ファイルダイアログの拡張子を.aveに変更する。
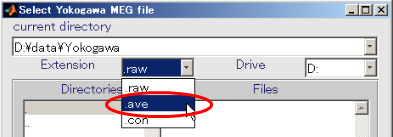
- Yokogawa MEGファイルを選択し、OKを押す(D:\data\Yokogawa\A008a_BC_0.5HPF_100LPF_trig437_label4.ave)
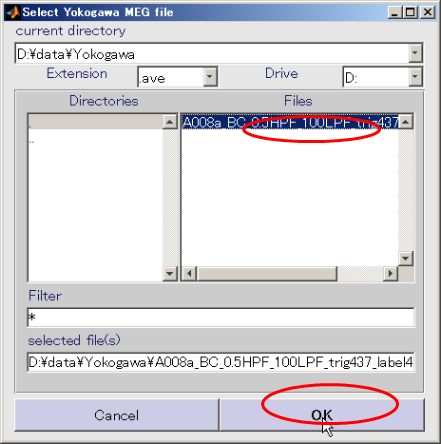
- Selectボタンを押し、センサ位置合わせファイルを選択する(D:\data\Yokogawa\marker1.pos.mat)。

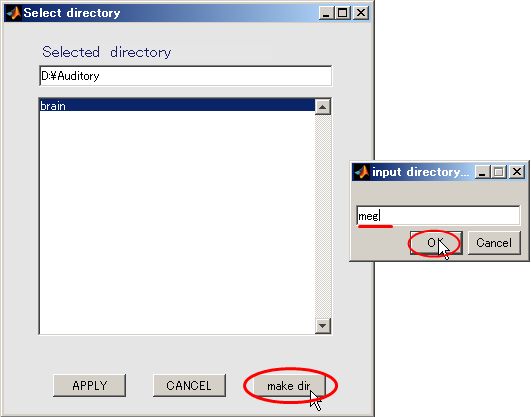
- 出力先を作成する(D:\Auditory\meg)
- Selectボタンを押す。

- make dirボタンを押すと、作成するディレクトリ名を入力するダイアログが表示されるので、megと入力し、OKボタンを押す。
- megを選択してから、APPLYを押す。
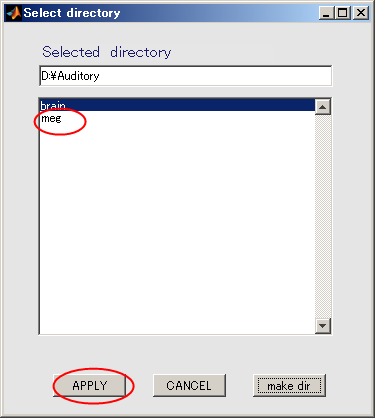
- 保存ファイル名が更新される。
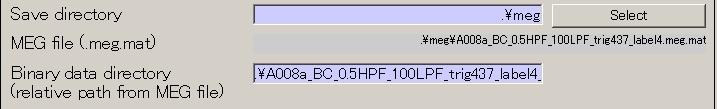
- Execボタンを押す。10秒程度で、MEGデータのインポートが完了する。
<<MATLAB Command window>>

リードフィールドを計算する
- [Analysis]->[Calculate leadfield]を選択する。
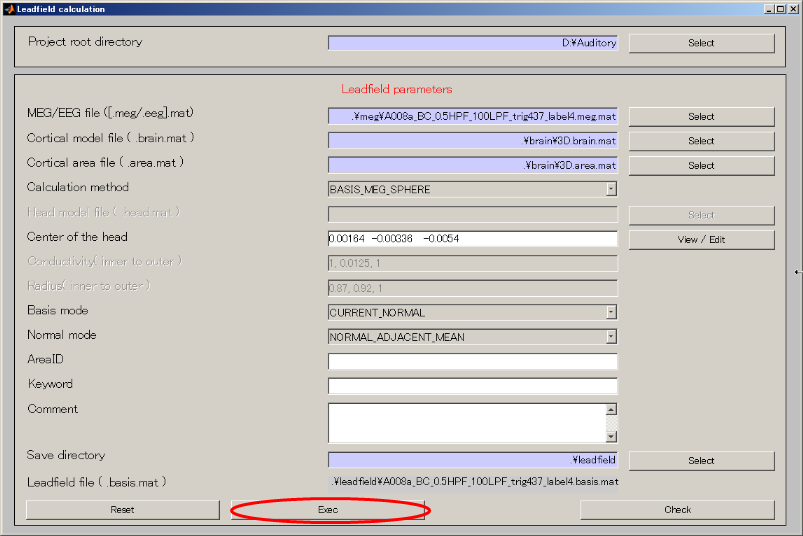
- MEG fileを指定する(D:\Auditory\meg\A008a_BC_0.5HPF_100LPF_trig437_label4.meg.mat)
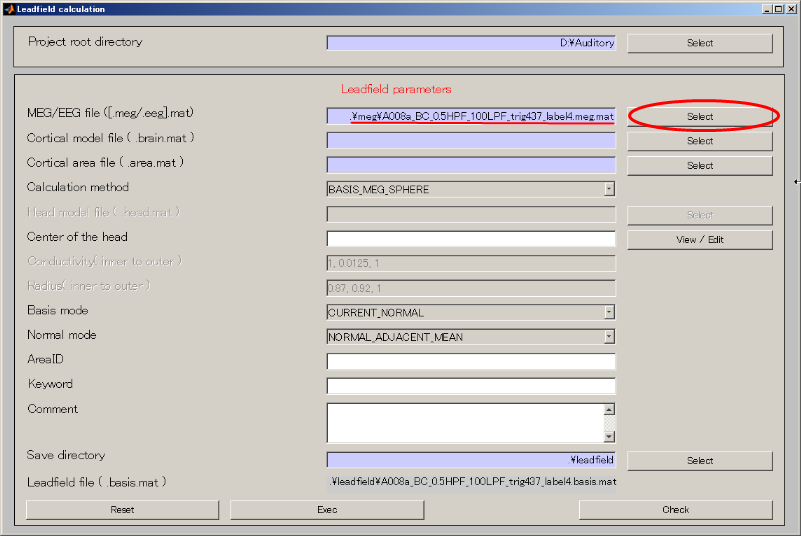
- Cortical model fileを指定する(D:\Auditory\brain\3D.brain.mat)。

- Cortical area fileを指定する(D:\Auditory\brain\3D.area.mat)。

- 出力先を作成する(D:\Auditory\leadfield)。
- Selectボタンを押す。

- make dirボタンを押すと、作成するディレクトリ名を入力するダイアログが表示されるので、leadfieldと入力し、OKボタンを押す。
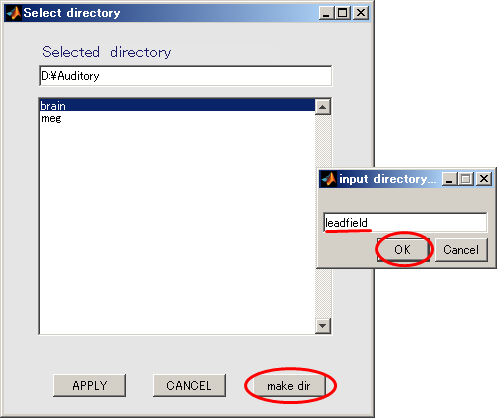
- leadfieldを選択してから、APPLYを押す。
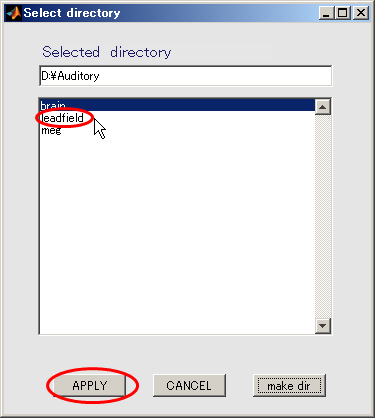
- 保存ファイル名が更新される。
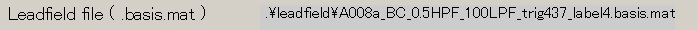
- Execボタンを押す。20~30秒程度で、リードフィールドの計算が完了する。
<<MATLAB Command window>>

電流分散を推定する
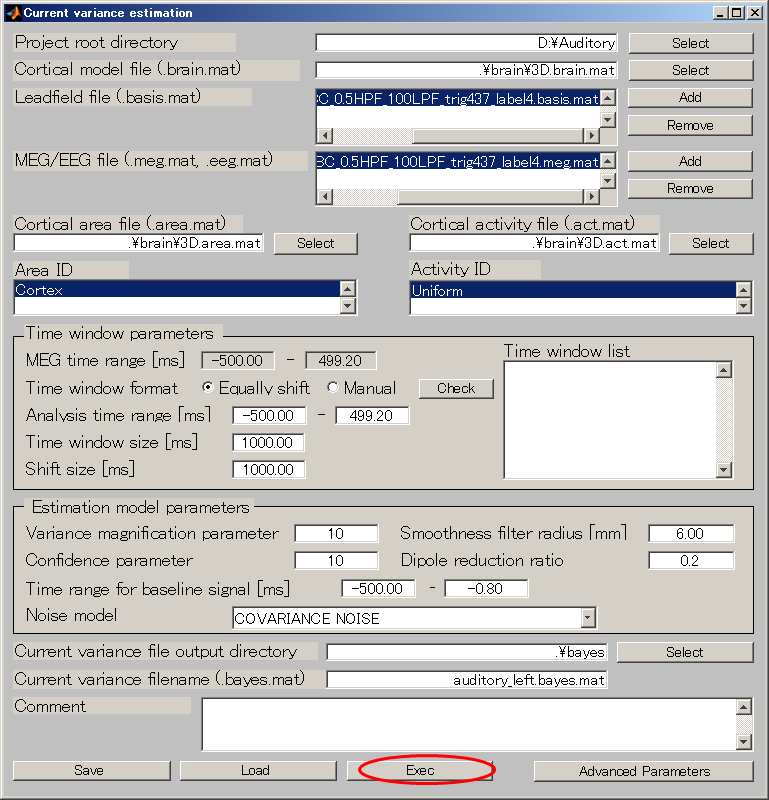
- [Analysis]->[Estimate current variance]を選択する。
- Cortical model fileを指定する(D:\Auditory\brain\3D.brain.mat)。
- Leadfield fileを指定する(D:\Auditory\leadfield\A008a_BC_0.5HPF_100LPF_trig437_label4.basis.mat)。

- MEG fileを指定する(D:\Auditory\meg\A008a_BC_0.5HPF_100LPF_trig437_label4.meg.mat)。

- Cortical area fileを指定する(D:\Auditory\brain\3D.area.mat)。
- Cortical activity fileを指定する(D:\Auditory\brain\3D.act.mat)。
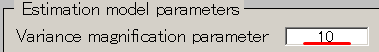
- Variance magnification parameterを10にする。
- Dipole reduction ratioを0.2にする。

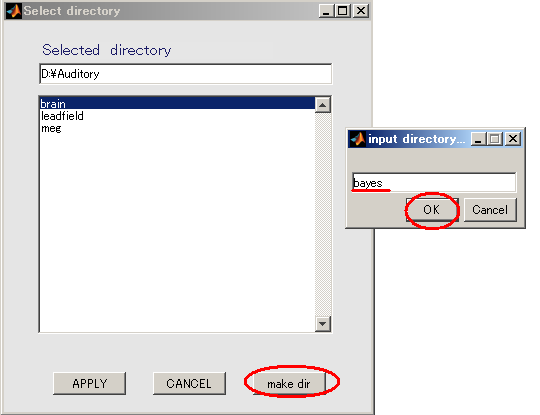
- 出力先を作成する(D:\Auditory\bayes)。
- Selectボタンを押す。

- make dirボタンを押すと、作成するディレクトリ名を入力するダイアログが表示されるので、bayesと入力し、OKボタンを押す。
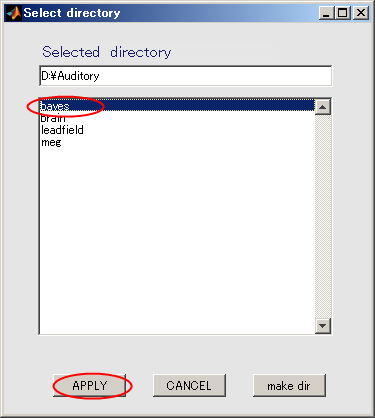
- bayesを選択してから、APPLYを押す。
- 保存ファイル名を入力する。
auditory_left[ENTER]で、拡張子: .bayes.matは自動的に付く

- Execボタンを押す。20分前後で推定が完了する。
<<MATLAB Command Window>>
Noise model: full covariance
Load MEG data for noise estimate [D:\Auditory\.\meg\A008a_BC_0.5HPF_100LPF_trig437_label4.meg.mat]
Session 1: -499.2 - 0.0[ms]
Number of basis files (= Number of sessions): 1
Area ID: Cortex
Number of vertices: 20004
--- Reduce cortex
Number of reduced vertices: 4004
--- Spatial smoothing filter calculation
R = 6.00e-003, Rmax= 1.20e-002
Number of vertices = 4004
Number of vertices in expanded area = 20002
Basis file for session 1: D:\Auditory\.\leadfield\A008a_BC_0.5HPF_100LPF_trig437_label4.basis.mat
Number of basis files (= Number of sessions): 1
Area ID: Cortex
Number of vertices: 20004
--- Reduce cortex
Number of reduced vertices: 4004
--- Spatial smoothing filter calculation
R = 6.00e-003, Rmax= 1.20e-002
Number of vertices = 4004
Number of vertices in expanded area = 20002
Basis file for session 1: D:\Auditory\.\leadfield\A008a_BC_0.5HPF_100LPF_trig437_label4.basis.mat
Number of sessions: 1
Time window: [1 1250]
MEG data file for session 1: D:\Auditory\.\meg\A008a_BC_0.5HPF_100LPF_trig437_label4.meg.mat
Number of sensors : 400
Number of trials : 1
--- Check variable consistency is OK
Noise model: spherical
Load MEG data for noise estimate [D:\Auditory\.\meg\A008a_BC_0.5HPF_100LPF_trig437_label4.meg.mat]
Session 1: -499.2 - 0.0[ms]
Number of sessions: 1
Time window: [1 625]
MEG data file for session 1: D:\Auditory\.\meg\A008a_BC_0.5HPF_100LPF_trig437_label4.meg.mat
Number of sensors : 400
Number of trials : 1
--- Initial VB-update iteration = 100
--- Total update iteration = 100
Sensor noise variance is estimated
Background current variance is estimated
Set prior fMRI activity pattern
--- New VBMEG estimation program ---
--- Initial VB-update iteration = 1000
--- Total update iteration = 1000
Tn= 1, Iter= 50, FE=16183629.600099, Error=5.920164e-002
Tn= 1, Iter= 100, FE=16184064.896437, Error=5.940531e-002
Tn= 1, Iter= 150, FE=16184112.004743, Error=5.944172e-002
Tn= 1, Iter= 200, FE=16184140.401725, Error=5.944495e-002
Tn= 1, Iter= 250, FE=16184143.926441, Error=5.944783e-002
Tn= 1, Iter= 300, FE=16184144.942685, Error=5.944801e-002
Tn= 1, Iter= 350, FE=16184145.363067, Error=5.944747e-002
Tn= 1, Iter= 400, FE=16184145.588821, Error=5.944667e-002
Tn= 1, Iter= 450, FE=16184145.749733, Error=5.944572e-002
Tn= 1, Iter= 500, FE=16184145.890986, Error=5.944465e-002
Tn= 1, Iter= 550, FE=16184146.025482, Error=5.944347e-002
Tn= 1, Iter= 600, FE=16184146.152109, Error=5.944222e-002
Tn= 1, Iter= 650, FE=16184146.263429, Error=5.944096e-002
Tn= 1, Iter= 700, FE=16184146.351531, Error=5.943978e-002
Tn= 1, Iter= 750, FE=16184146.413128, Error=5.943876e-002
Tn= 1, Iter= 800, FE=16184146.451154, Error=5.943794e-002
Tn= 1, Iter= 850, FE=16184146.472196, Error=5.943733e-002
Tn= 1, Iter= 900, FE=16184146.482881, Error=5.943690e-002
Tn= 1, Iter= 950, FE=16184146.487982, Error=5.943661e-002
Tn= 1, Iter=1000, FE=16184146.490319, Error=5.943642e-002
Alpha is scaled back by bsnorm
----- Save estimation result in D:\Auditory\.\bayes\auditory_left.bayes.mat
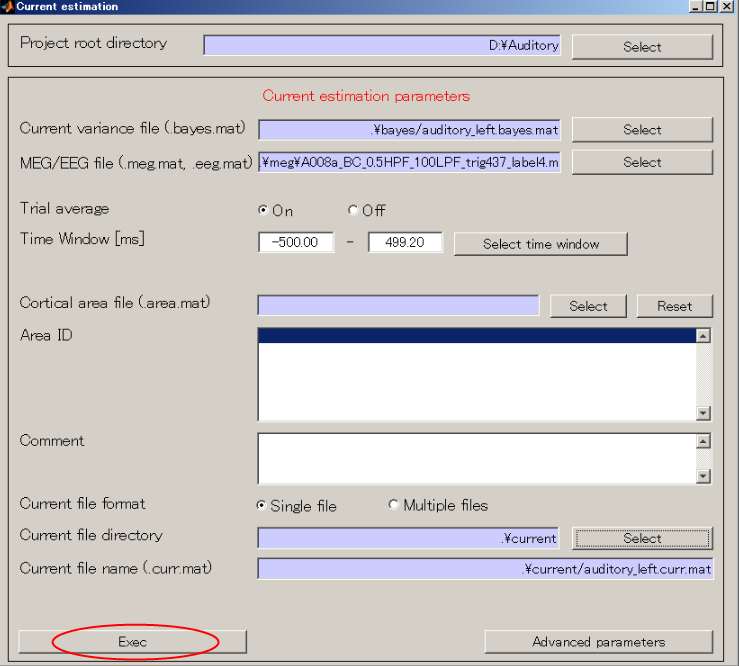
電流を計算する
- [Analysis]->[Estimate current]を選択する。
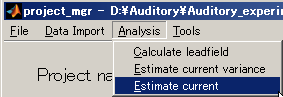
- Current variance fileを指定する(D:\Auditory\bayes\auditory_left.bayes.mat)。
自動的にMEG fileはセットされる。
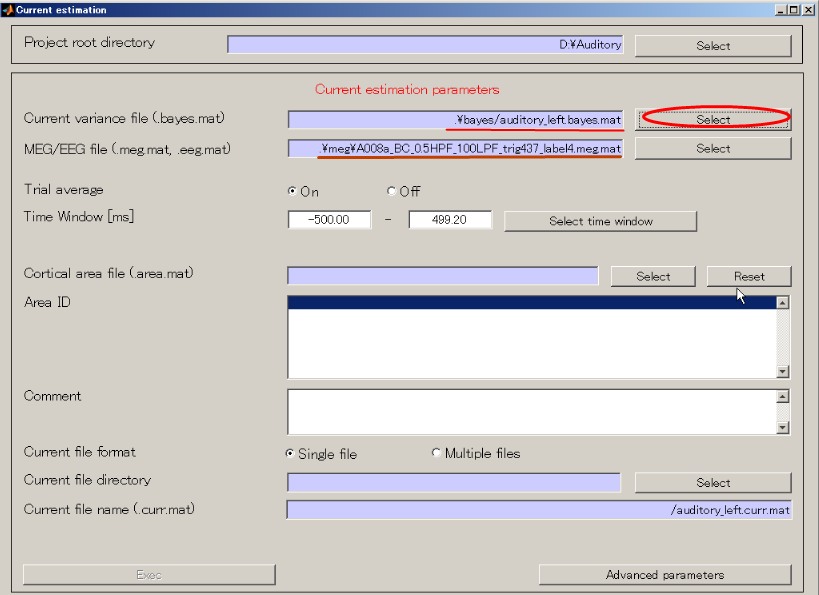
- 出力先を作成する(D:\Auditory\current)。
- Selectボタンを押す。

- make dirボタンを押すと、作成するディレクトリ名を入力するダイアログが表示されるので、currentと入力し、OKボタンを押す。
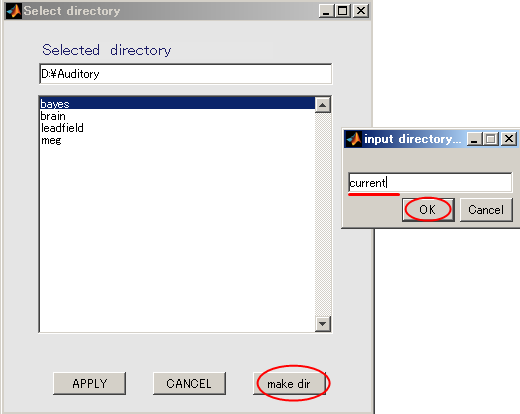
- currentを選択してから、APPLYを押す。
- 保存ファイル名が更新される。
- Execボタンを押して、1分程度で電流計算が完了する。
<<MATLAB Command Window>>
----- New VBMEG -----
Start current estimation
Number of sessions: 1
Time window: [1 1250]
MEG data file for session 1: D:\Auditory\.\meg\A008a_BC_0.5HPF_100LPF_trig437_label4.meg.mat
Number of sensors : 400
Number of trials : 1
--- Lead field matrix of focal window
Number of basis files (= Number of sessions): 1
Area ID: Cortex
Number of vertices: 20004
--- Reduce cortex
Number of reduced vertices: 4004
--- Spatial smoothing filter calculation
R = 6.00e-003, Rmax= 1.20e-002
Number of vertices = 4004
Number of vertices in expanded area = 20002
Basis file for session 1: D:\Auditory\.\leadfield\A008a_BC_0.5HPF_100LPF_trig437_label4.basis.mat
--- Save estimated current: D:\Auditory\.\current/auditory_left.curr.mat
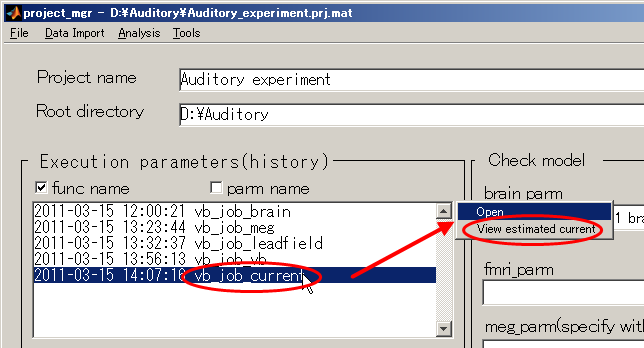
推定電流を見る
- project_mgr GUIを表示し、vb_job_currentを選択する。
すると、右上にポップアップメニューが表示されるので、View estimated currentを選択する。
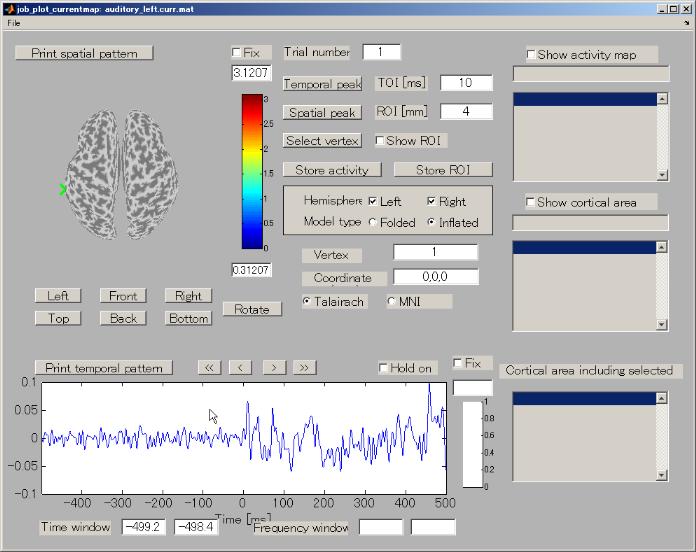
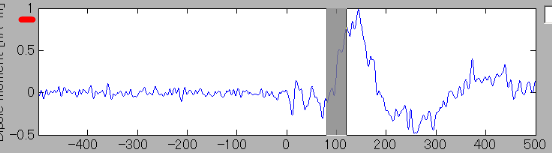
- 推定電流を表示するためのGUIが表示される。画面左上の空間分布表示パネルには、選択されている時間窓(time of interest;
TOI)で平均された電流強度分布が皮質モデル(inflated
model)上に表示される。画面下部の電流時系列表示パネルには、選択されている関心領域(region of interest;
ROI)に含まれる頂点に渡り平均された電流強度時系列が表示される。ROIのサイズはデフォルトで半径4mmである。
- TOIは"Time window"エディットテキストで設定できる。ここではTOIを、左右両側に表れる聴覚関連磁場(AEF)の潜時である、80~120msecに設定する。
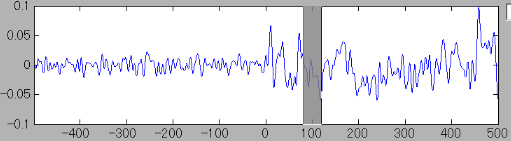
TOIの設定に伴い、空間分布表示パネル上の電流強度空間分布が更新される。TOIは時系列表示パネル上で次のように灰色で表示される。
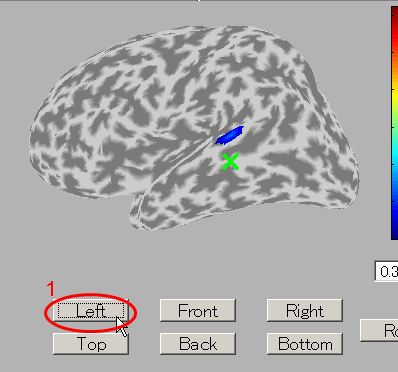
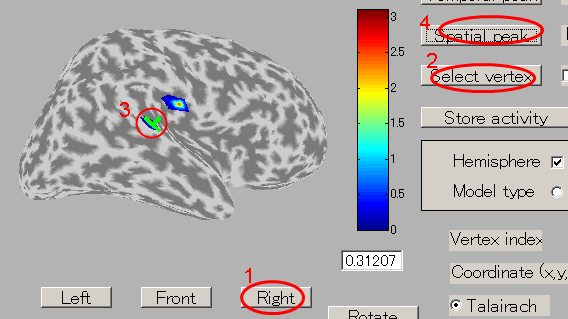
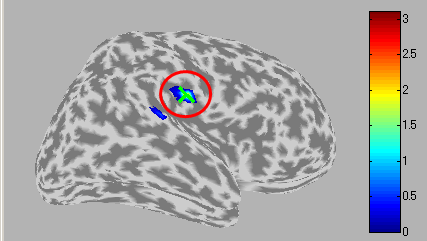
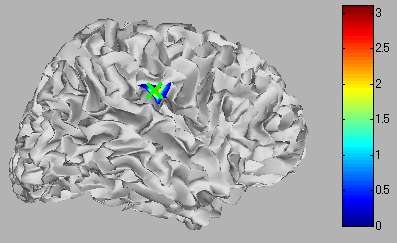
- Leftボタンを押すと皮質モデルを左側から見ることができる。聴覚野付近に脳活動が見られる。
- ROIの中心を選択するために、"Select vertex"ボタンを押してから皮質モデル上の1点を選択する。ここでは左脳聴覚野付近の脳活動源を選択する。
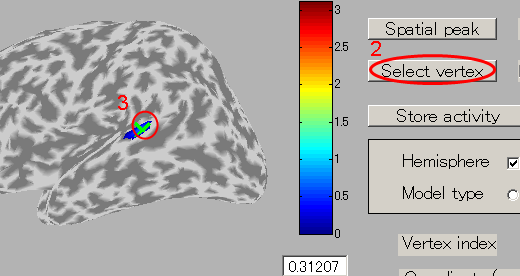
"Vertex index"エディットボックスに選択された頂点のインデックスが表示される。

このエディットボックスに頂点番号を直接入力する事でもROIの中心を変更できる。
ROIの中心は皮質モデル上で緑の×印で示される。電流時系列表示パネルには選択されたROIの電流時系列が表示される。Y軸のスケールは電流時系列に合わせて変わる。
- より電流強度の高い頂点を探し出すために、"Spatial peak"ボタンを押す。するとROIの中で電流強度の時間平均が最大の頂点が探索され、ROIの中心がその頂点に設定される。
同時に"Vertex index"エディットボックスの頂点番号が更新される。

電流時系列表示パネルに更新されたROIの電流時系列が表示される。
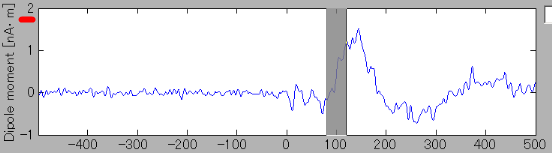
- 同様の手順で右脳聴覚野付近の推定電流を見ることができる。
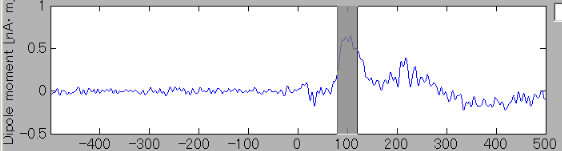
推定電流を見る(補足)
- 右脳聴覚野付近の脳活動源の前方に、もう一つ脳活動源が見られる。
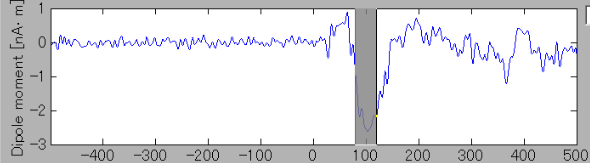
-
これら二つの脳活動源の時系列を比較すると、一方で電流が内向き(時系列表示上で正)のとき、もう一方では外向き(負)になっている。これは、一つの脳活
動源が二つの脳活動源に分離されたものと考えられる。このような推定脳活動源の分離は、二つの脳活動源(頂点)のリードフィールドが互いに反転したパター
ンを持つときに起こり得る。リードフィールドの吸い込みと湧き出しの空間位置は頂点位置によって決まるので、二つの頂点は互いに近い位置にあると予想され
る。さらに、リードフィールドの正負、すなわち吸い込みと湧き出しの向きは頂点位置での皮質法線方向で決まるので、二つの頂点位置ではそれらの法線方向が
互いに逆向きになっていると予想される。
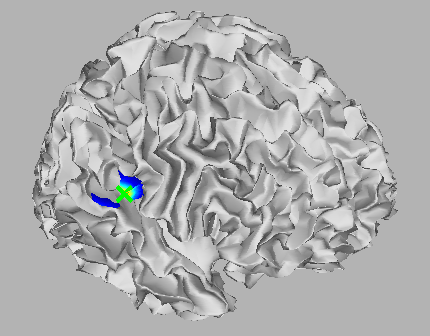
二つの要因のうち、頂点位置についてGUI上で定性的に確認する。"Inflated model"表示では元の皮質モデル(白質/灰白質境界)の位相情報しか保たれていない。元の皮質モデルを見るために、表示を"Folded model"に変更する。
すると元の皮質モデル上が表示される。
右脳聴覚野付近の脳活動源は皺に隠れている。表示角度を変える事でこの脳活動を見ることができる。
- リードフィールドを決定する二つの要因である頂点位置と法線方向について定量的に調べるため、MATLABコンソール上で作業を行う。次のコマンドによって、頂点位置と法線方向が変数"V"と"xx"にそれぞれ代入される。
>> [V,F,xx] = vb_load_cortex('3D.brain.mat'); - 二つの頂点間の距離と法線ベクトル方向余弦は次のようにして得られる。
sqrt(sum((V(19805,:)-V(19617,:)).^2)) ans = 0.0141 >> sum(xx(19805,:).*xx(19617,:)) ans = -0.7452 >>以上の通り、頂点間距離は14.1cm、方向余弦は-0.745であった。これより二つの脳活動源に対応するリードフィールドが互いに反転したパターンを持つ事が示唆される。